Contrôler la bioproduction de médicaments en temps réel grâce à la microscopie in situ et l'intelligence artificielle
Focus sur un outil développé à l'Institut Pascal qui permettra de mieux contrôler les procédés de fabrication de médicaments biopharmaceutiques, et qui ouvre la voie à de nouvelles recherches. Un projet interdisciplinaire de l'IdS (Institut Pascal, Axe ISPR) et de Clermont Auvergne INP (Institut Pascal, Axe GePEB).
Republication de l'article disponible sur le site du CNRS Ingénierie, Contrôler la bioproduction de médicaments en temps réel grâce à la microscopie in situ et l'intelligence artificielle | CNRS Ingénierie
La classification des cellules dans un bioréacteur est réalisée par un réseau de neurones qui analyse en temps réel les images captées par un microscope in situ. Cet outil développé à l'Institut Pascal permettra de mieux contrôler les procédés de fabrication de médicaments biopharmaceutiques, et ouvre la voie à de nouvelles recherches. Ces résultats sont publiés dans Nature Scientific Reports.
La production de médicaments biopharmaceutiques (anticorps, protéines...) est réalisée dans des bioréacteurs contenant des cellules vivantes. De nombreux paramètres du procédé, tels que la concentration en oxygène, le pH, la présence de nutriments ou de sous-produits, sont susceptibles d'altérer le fonctionnement et la viabilité de ces cellules. Mais aucun instrument, jusqu'à présent, ne permettait d'analyser finement et en temps réel l'état des cellules au sein d'un bioréacteur. En combinant la microscopie in situ et l'intelligence artificielle, des scientifiques de l'Institut Pascal (CNRS/Université Clermont Auvergne) ont mis au point un outil qui réalise en temps réel une classification des cellules dans le réacteur, des cellules parfaitement viables jusqu'aux cellules mortes.
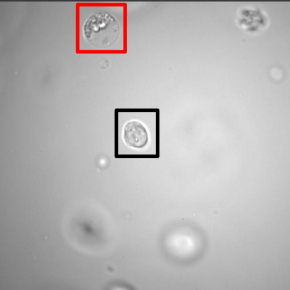
Le développement du nouvel instrument a démarré par l'examen d'une base de 4000 images prises par microscopie in situ, qui a permis de définir six classes de cellules en fonction de leur morphologie, caractéristique de leur état : viable, morte, ou en transition réversible ou non entre les deux. Plus de 900 cellules appartenant aux différentes classes ont ainsi été ''étiquetées''. L'apprentissage d'un algorithme de détection de cellules, basé sur un réseau de neurones artificiels, a ensuite été réalisé sur un ensemble de 3400 images, le reste – 600 images- étant réservé pour la phase de tests. Une fois l'algorithme optimisé, ces tests de performance ont montré que l'algorithme classe les cellules avec un taux de réussite qui peut atteindre 95%. Afin de réaliser une classification en temps réel, condition indispensable pour une application en contrôle de procédé d'un bioréacteur, les scientifiques ont utilisé un réseau de neurones capable de livrer un résultat en quelques millisecondes.
Pour effectuer un test dans des conditions représentatives, le nouvel outil a été connecté à un une cuve contenant une culture de cellules produisant des anticorps monoclonaux. Dans ce réacteur de laboratoire, extrapolable à l'échelle industrielle, des variations brutales de paramètres (O2, pH, nutriments...) ont été volontairement créées, afin d'induire différentes voies de mort cellulaire. Près de 850.000 images ont ainsi été analysées par le dispositif de classification de cellules. Dans le cadre d’un procédé industriel, le nouvel instrument devrait permettre de détecter de façon précoce l’apparition des processus de mort cellulaire, afin d’en limiter l’effet en modifiant les paramètres du procédé.
Dans la continuité de cette étude, un nouveau projet a été démarré, avec deux partenaires, afin d'étendre les performances du système à d'autres types de cellules et bioprocédés. Par ailleurs, la capacité inédite d'observer et de classer rapidement des centaines de milliers de cellules ouvre de nombreuses possibilités de recherche sur les différentes voies de mort cellulaire en bioréacteur.
Ci-dessous : Schéma du processus de mise au point de l'outil de classification de cellules.
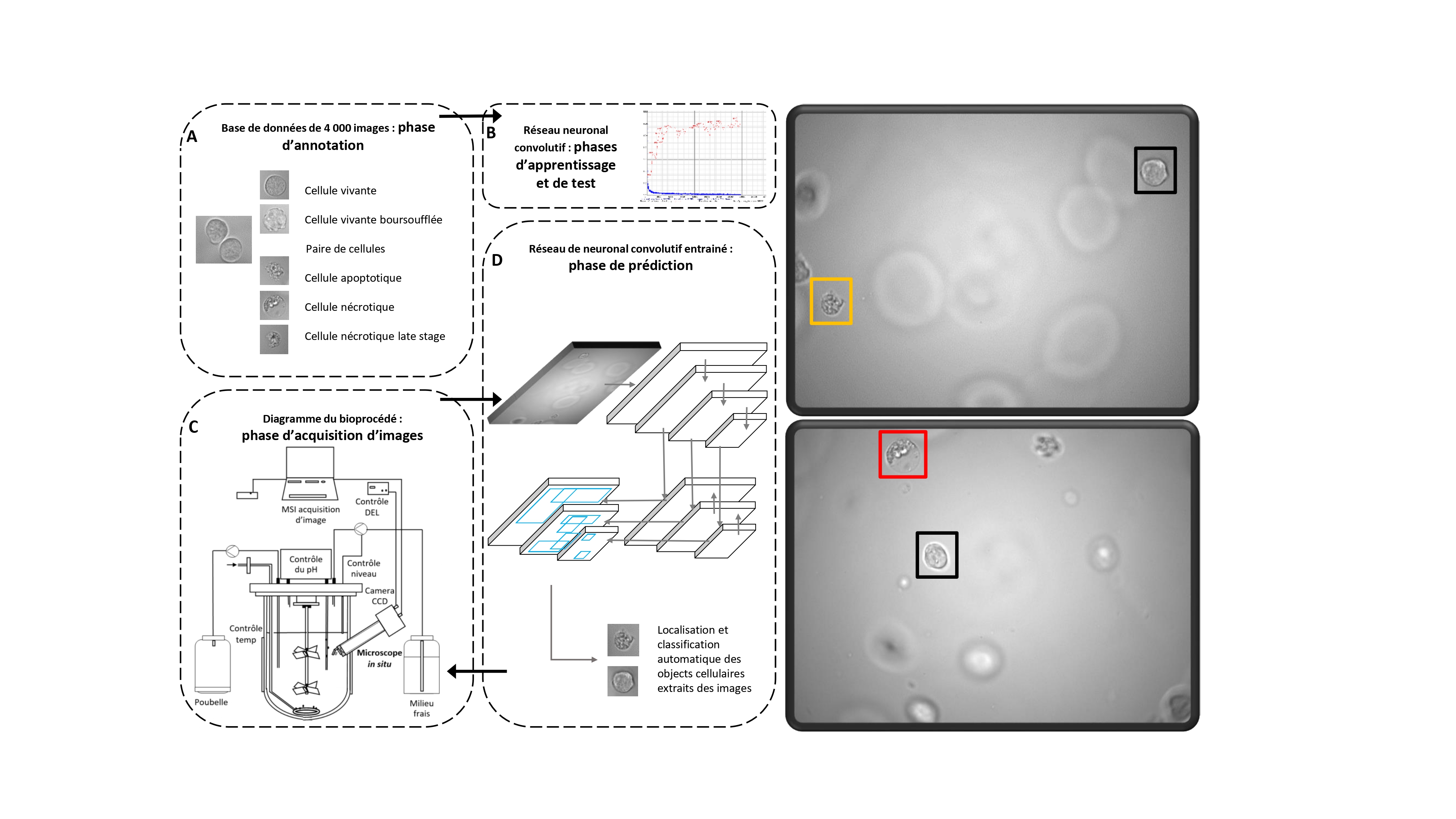
Ci-dessous : Six classes de cellules sont identifiées par leur morphologie.

Publication : Deep in situ microscopy for real-time analysis of mammalian cell populations in bioreactors.
Auteurs : Jean-Sébastien Guez, Pierre-Yves Lacroix, Thierry Château & Christophe Vial.
Nature Scientific Reports, publié le 12 décembre 2023.
https://doi.org/10.1038/s41598-023-48733-x
Article consultable sur la base d’archives ouvertes HAL
